Investigadores del Instituto de Investigación Biológica (IRB) de Barcelona han creado «Bioteque», una herramienta computacional capaz de almacenar y relacionar millones de datos sobre genes, enfermedades o células y que permite unificar los datos biológicos disponibles
La creación de la «Bioteque» se basa en algoritmos de inteligencia artificial para generar descriptores.
El jefe del laboratorio de Bioinformática Estructural y Biología de Redes del IRB, Patrick Aloy, ha explicado que la rápida evolución de las distintas disciplinas en los campos de la investigación biológica y biomédica, ha hecho que en las últimas décadas la cantidad de datos biológicos disponible haya crecido de manera exponencial.
Por ejemplo, en el Instituto Europeo de Bioinformática (EMBL-EBI) han pasado de gestionar y almacenar un volumen de 40 petabytes a trabajar con 250 petabytes, en tan solo 6 años.
Por eso, los científicos del IRB, han desarrollado esta herramienta computacional «para armonizar, integrar y simplificar estos datos».
Más de 30 millones de interacciones funcionales
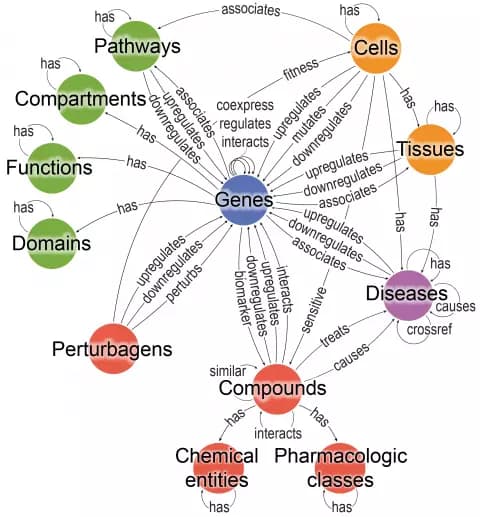
Se trata de un «panel de conocimiento» que proporciona información sobre cómo las distintas entidades biológicas se relacionan entre sí. Incluye más de 30 millones de interacciones funcionales.
La «Bioteque» funciona integrando distintos niveles de complejidad biológica. Puede reportar, por ejemplo, sobre dos genes que están relacionados, si interaccionan físicamente, si son activos en el mismo tipo de células, si están relacionados con la misma enfermedad, o predecir la sensibilidad o resistencia de un tipo de células ante un fármaco concreto.
“Este recurso computacional que hemos desarrollado es de los primeros dirigidos a unificar informaciones biológicas y el único en abordar tal diversidad y cantidad de datos. Permite acceder, de forma fácil y armonizada a prácticamente todo el conocimiento biológico disponible a día de hoy. Tiene un potencial enorme para acelerar la investigación biomédica», ha destacado Aloy.
Han trabajado con 150 bases de datos
En la «Bioteque» la información está estructurada en 12 tipos de entidades biológicas como: gen, enfermedad, tejido, célula…
Para cada una de estas entidades, contempla una serie de descriptores o características, por ejemplo: el patrón de mutaciones de un gen, el perfil de interacciones físicas de las proteínas resultantes, la expresión de dicho gen en distintos tipos celulares o su relación con enfermedades.
«Hemos trabajado con información procedente de 150 bases de datos distintas. Primero tuvimos que ponerlas todas en el mismo lenguaje. Y luego convertir ese conocimiento en descriptores numéricos que pudieran ser interpretados por los algoritmos. Para así poder explotar computacionalmente esas redes y conexiones», ha detallado Adrià Fernández, estudiante de doctorado en el laboratorio del IRB.
La «Bioteque», que se irá ampliando periódicamente con nuevas bases de datos, según se vayan haciendo públicas, es de acceso abierto en https://bioteque.irbbarcelona.org/.


